|
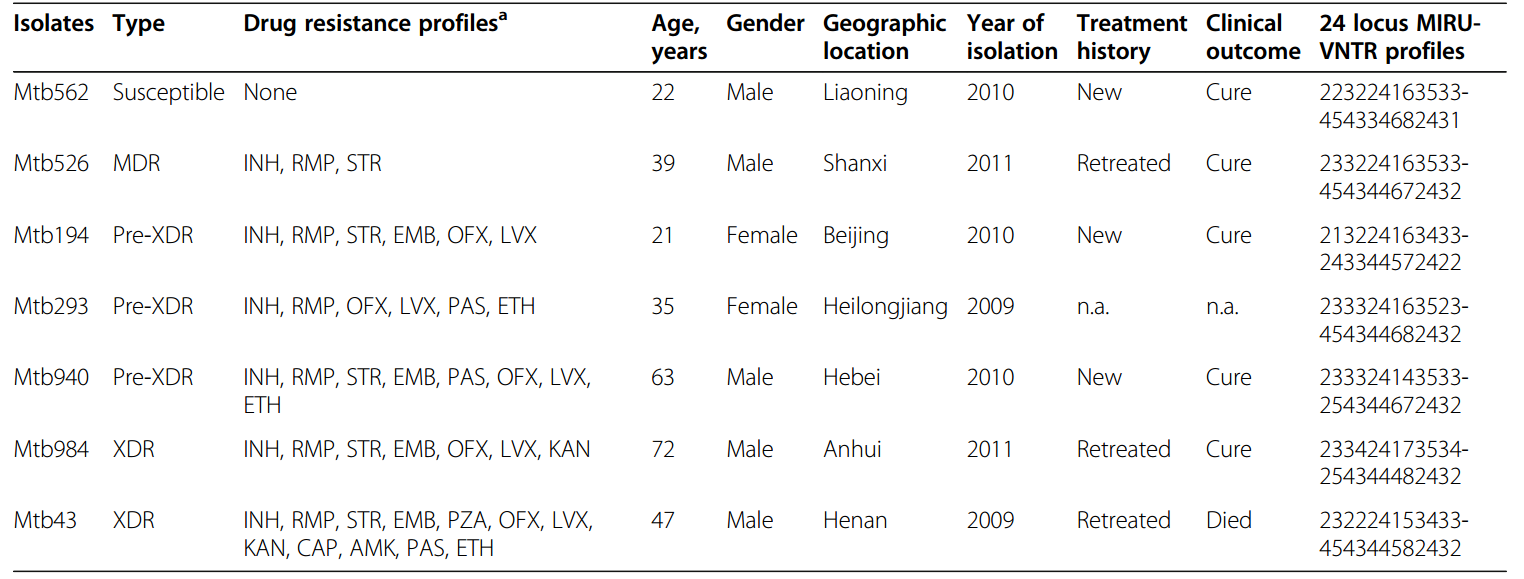
英文题目:Comparative genomic analysis of Mycobacterium tuberculosis clinical isolates 中文题目:结核分支杆菌临床分离菌株的比较基因组学分析 期刊名: BMC Genomics 发表时间:2014,6 IF:3.867 技术: Whole genome sequencing(PE100) 测序平台:Illumina 测序平台 材料:7株结核分枝杆菌临床分离菌株 研究背景: 由于过多抗生素的使用,抗药性的结核分枝杆菌已成为许多国家严重威胁公共卫生和疾病控制的主要障碍。为了更好了解抗药性结核分枝杆菌菌株的进化,研究者对7株具有不同抗药性范围的结核分支杆菌分离株进行全基因组测序,且通过比较基因组学分析基因变异性。 分析方法:SOAP denovo组装/COG/KEGG功能注释/MEGA5进化树构建 研究结果: 全基因组序列统计 表1是7株临床分离菌株信息,包括抗药类型,年龄,抗药图谱,分离时间等相关信息。每个菌株基因组序列覆盖度在200-560×,完整度在97.43-97.82%。通过与参考临床分离菌株H37Rv基因组序列进行对比,发现7株菌株的抗药图谱不同,且有着相似的SNPs(数量范围从1409-1464)和Indels(56-101)。
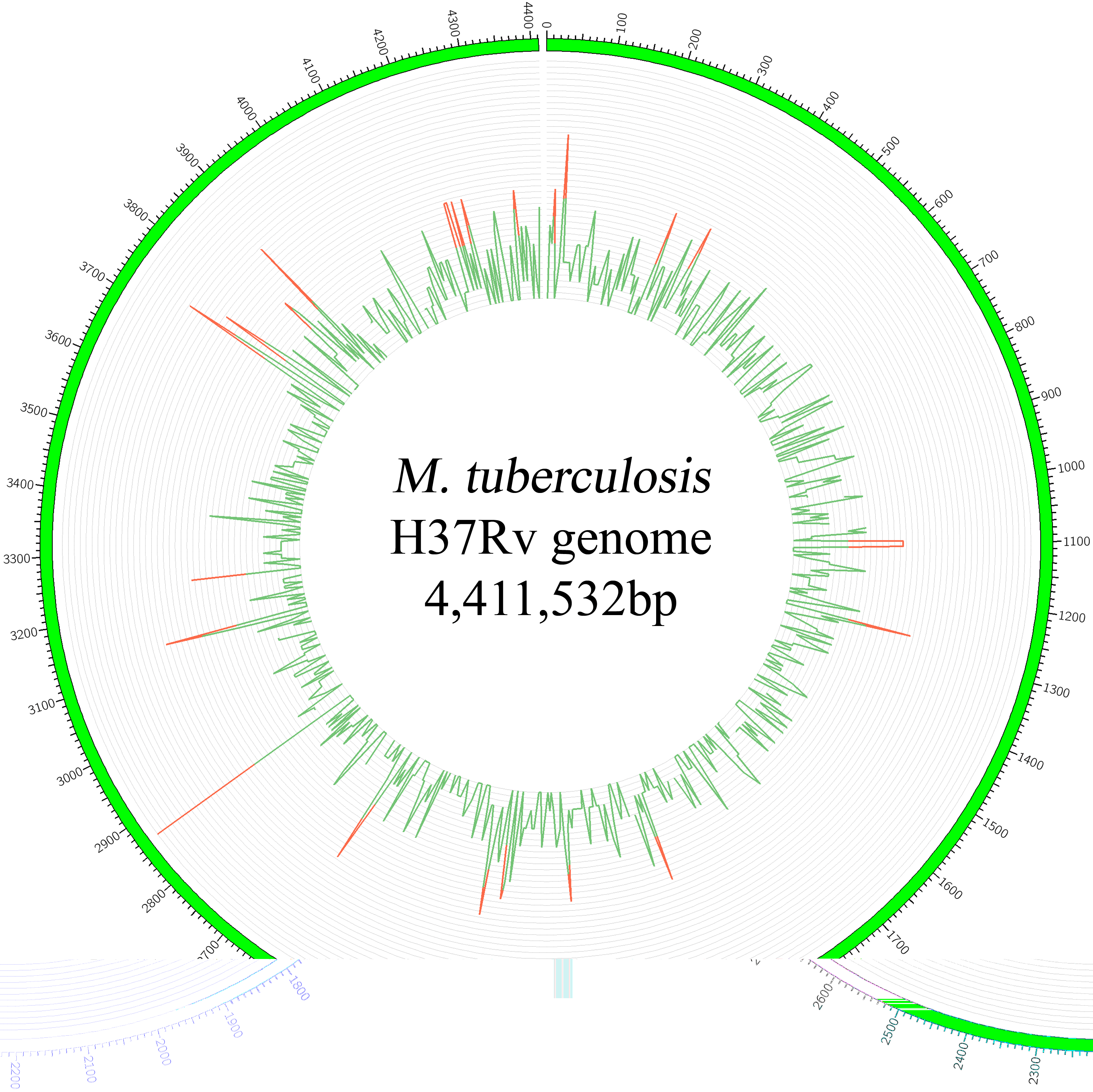
表1:临床结核分枝杆菌的流行病学和临床资料。INH, 异烟肼; RMP, 利福平; STR, 链霉素; EMB,乙胺丁醇; PZA,吡嗪酰胺; OFX,氧氟沙星; LVX, 左氧氟沙星; KAN, 卡那徽素; CAP,卷曲霉素;AMK, 氨基羟丁基卡那霉素A; PAS, 对氨基水杨酸; ETH, 硫异烟胺. 结核分支杆菌基因组中SNP聚类和分布 通过进一步的基因组学比较分析,总共有1871个非重复性SNPs,其中7株菌株共有的有1102个SNPs。SNP密度图显示了SNPs的非随机分布,有25个区域有较高的SNP密度(图1中的红色条带)。通过进一步不同等级的COG注释进一步分析了SNP的分布,结果发现较少SNPs富集在于次级代谢生物合成,运输和代谢相关基因上(Class Q),较多SNP富集在一些未知功能基因上(Class S),细胞壁/膜合成(Class M),通用功能(Class M),防御机制(Class V),翻译,核糖体结构和合成(Class J),转录(Class K),信号转导(Class T),细胞移动(Class N)。
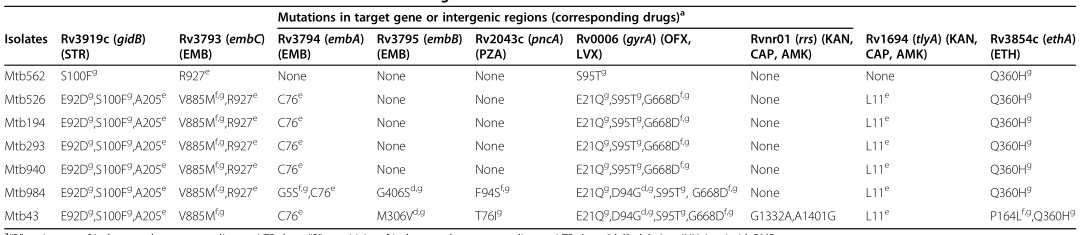
图1 SNP密度Circos分布图。绿色带表示非重叠5K区段内的SNP密度;红色带表示具有显著SNP高密度的区域。 染色体组的插入和缺失 和H37Rv菌株进行对比发现本研究中的菌株有大片段插入和缺失(插入或者缺失片段至少20bp长度),共发现了并发现了1个菌种非特异性和29个菌种特异性大片段插入,2个菌种非特异性和61个菌种特异性大片段缺失。并在抗原变异主要来源PE-PPE-PGRS基因上发现了16处Indel。许多Indels在对药物敏感和抗药性菌株中被确定。 结核分支杆菌基因组中CRISPR的分布 使用CRISPRfinder去推测7株结核分支杆菌菌株基因组中的CRISPR位点。与已预测出有两个CRISPR结构的实验室分离株H37Rv进行比较,发现本研究中的7株临床分离菌株和先前测序的其他两株临床结核分枝杆菌菌株(CCDC5079,CCDC5180)只有这两个CRISPR结构中的一个。且证实抗生素的耐药性和CRISPR-case区之间没有相关性。 结核分支杆菌中与抗药性相关的基因突变 通过分析比较发现7株中国临床分离菌株中共有39种类型的突变与抗药性相关,包括14个先前已经报道的,以及25个新鉴定出的位点。抗药表型和突变之间的关联在不同药物之间变化很大(例如isoniazid的抗药性相关位点在不同菌种之间完全相同,而para-aminosalicylic acid的抗药性相关位点在不同菌种之间完全不同)。7株中国临床分离菌株中与抗药性相关的突变请见表2。
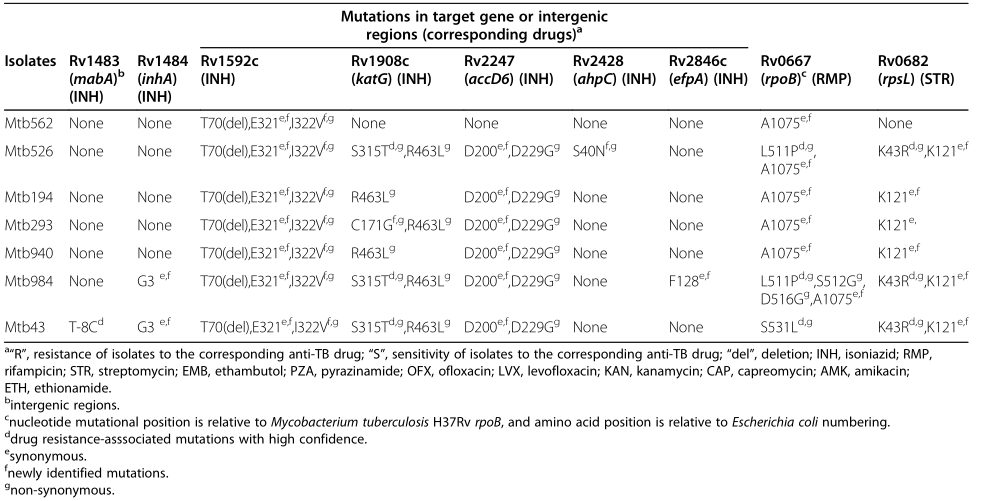
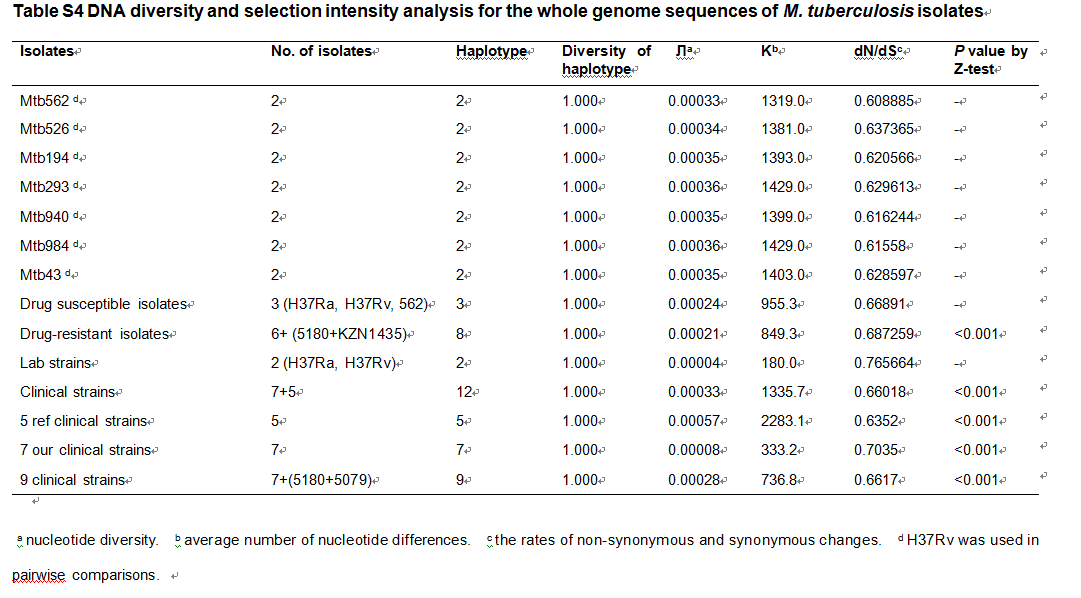
表2:7株中国临床分离菌株中与抗药性相关的SNP位点和插入 结核分支杆菌基因组的遗传多样性和选择强度 使用结核分支杆菌全基因组序列进行遗传多样性和选择强度分析,结果显示这7个新的临床分离菌株有着相似的核苷酸多样性(Л值),分布范围在0.00033到0.00036,在对药敏感和抗药菌株之间核苷酸多样性无显著性差异(0.00024 vs. 0.00021),而在临床分离菌株和实验室分离菌株之间则有显著性差异(0.00033 vs. 0.00004)。这7个新的临床分离菌株和5个以前描述过的临床分离菌株之间的Л值有明显区别(0.00008 vs. 0.00057)。 全基因组序列的dN/dS值在具有不同抗药图谱的分离菌株之间很相似,数值分布在0.608885-0.637365之间,在对药敏感和抗药菌株间无显著性差异,而临床分离菌株的dN/dS值显著低于实验室分离菌株(0.66018 vs. 0.765664)。
表3:结核分支杆菌全基因组序列的遗传多样性(Л值)和选择强度分析(dN/dS) 结核分支杆菌菌株的系统发育树分析 采用邻接法(NJ)和最大相似性(ML)两种方法对7株菌株和其他7株结核分支杆菌全基因组中的SNPs进行菌株的系统发育树分析,结果显示不同的临床分离菌株的进化关系在两种进化树中是相似的。这7株新的结核分支杆菌临床分离株与和之前北京地区菌株CCDC5079和CCDC5180在进化树中形成一个分支,表明进化亲缘关系较近。
图2 基于全基因组中的SNPs菌株间的进化关系 研究结果: • 结果发现7株不同抗药图谱的临床分离菌株有着相似数目的SNPs,数量范围从1409-1464。大片段的插入和缺失(Indels)数量也很相似(56-101)。 • 通过对抗性数据库注释发现39处与抗药性相关的变异,包括14处先前报道的位点以及25处新的位点,并在抗原变异主要来源PE-PPE-PGRS基因上发现了16处Indel。 • 发现了一个在7株临床分离菌株上具有不同间隔的CRISPR簇,表示它在结核分支杆菌基因组可塑性上发挥着重要作用。 • 7株临床分离菌株基因组的核苷酸多样性((Л值)和选择强度(dN/dS值)非常相似,所有7株临床分离菌株的dN/dS值都小于1(范围从0.608885到0.637365),这表明结核分枝杆菌基因组正在经历净化选择作用。药物敏感菌株和抗药性菌株的核苷酸多样性((Л值)和选择强度(dN/dS值)是相似的。 总体评价: 本研究采用全基因组测序,揭示了临床结核分支杆菌分离菌株在遗传组成上有一定的相似性,而在SNP的分布,Indels,CRISPR-cas结构及核苷酸多样性,选择强度上有明显的变化。通过COG功能注释显示药物敏感菌株和抗药性菌株在基因组水平上并没有显著的差异,表明临床分离菌株的抗药性进化过程是十分复杂而多变的。除此之外,研究还需要对几个问题进行深入的调查,如菌株的药物敏感度是否受特定基因的缺失和特定代谢途径的影响,选用更多的来自于不同分支类群的结核分支杆菌菌株更好地了解耐药性结核分支杆菌的进化演变等等。 |